2019年5月26日 讯 /基因宝jiyinbao.com/ –本期为大家带来的是基因编辑领域的最新研究进展,希望读者朋友们能够喜欢。
1. Science子刊:一种新型腺嘌呤碱基编辑器可让细胞RNA编辑最小化
DOI: 10.1126/sciadv.aax5717.
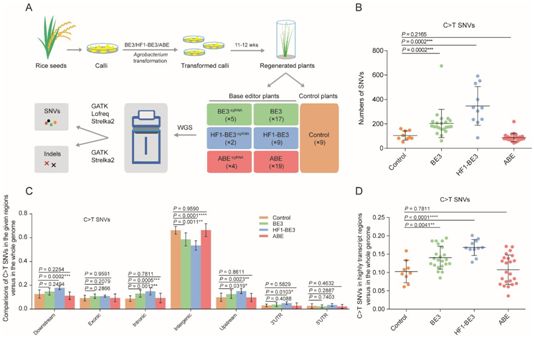
在一项新的研究中,来自美国布罗德研究所和哈佛大学的研究人员发现有证据表明使用碱基编辑器会导致细胞中出现意想不到的RNA编辑。相关研究结果发表在2019年5月8日的Science Advances期刊上,论文标题为“Analysis and minimization of cellular RNA editing by DNA adenine base editors”。在这篇论文中,他们描述了他们对CRISPR类型腺嘌呤碱基编辑器(ABE)的研究,以及他们取得的发现。
ABE将一个DNA碱基对转换成另一个DNA碱基对,从而允许修复某些细胞类型中的突变,而不会产生不想要的编辑效应。据认为,ABE还有潜力校正几乎一半已知的导致医学疾病的遗传异常。ABE的科学基础对医学界来说变得越来越重要。不幸的是,最近的一些研究已发现,ABE可能也会导致意料之外的编辑。在今年3月,一个研究团队发现胞嘧啶碱基编辑器3型(CBE3)以高于正常的速率诱导单核苷酸变异。在上个月,另一个研究团队发现胞嘧啶碱基编辑器(CBE)和ABE导致RNA中的脱靶编辑。在这项新的研究中,这些研究人员试图在使用ABE时进一步测试脱靶编辑,并在确认后找到一种解决方案。
这些研究人员以一种包括人细胞系中所有细胞RNA转录本的方式分析了ABE的最新版本,称为ABEmax,而且他们使用比其他人使用的更灵敏的工具来做到这一点。他们报道他们确实在RNA样本中发现了低水平的脱靶编辑。为了解决这个问题,他们开发了在保留在靶碱基编辑的能力同时导致较少RNA编辑的新型ABE变体(基于灭活的野生型大肠杆菌)。他们进一步报道说,这些新型变体是以一种让RNA和DNA编辑过程解耦合的方式构建出来的,这样就能够最大限度地减少DNA和RNA中的脱靶编辑。
这些研究人员得出结论:由于较低的RNA编辑且较短的RNA半衰期,对未来研究的干扰程度可能取决于它们的具体应用。他们建议寻求让RNA编辑最小化的科学家们使用他们构建出的一种新变体—他们称之为ABEmaxQW。
2. Cell:首次发现阻断CRISPR-Cas9基因组编辑的小分子抑制剂
doi:10.1016/j.cell.2019.04.009.
在一项新的研究中,来自美国布罗德研究所等研究机构的研究人员发现酿脓链球菌Cas9(SpCas9)的首批小分子抑制剂能够更精确地控制基于CRISPR-Cas9的基因组编辑。具体而言,他们通过开发一系列高通量生物化学分析方法和基于细胞的分析方法,筛选了许多小分子,以便鉴定出能够破坏SpCas9与DNA结合因而干扰它的DNA切割能力的化合物。这些首批小分子CRISPR-Cas9抑制剂很容易进入细胞,并且比之前发现的抗CRISPR蛋白小得多。这些新化合物可以对基于SpCas9的编辑技术进行可逆的和剂量依赖性的控制,包括它们在哺乳动物细胞中进行基因编辑、碱基编辑和表观遗传编辑的应用。相关研究结果发表在2019年5月2日的Cell期刊上,论文标题为“A High-Throughput Platform to Identify Small-Molecule Inhibitors of CRISPR-Cas9”。
论文通讯作者、布罗德研究所的Amit Choudhary说道,“这些技术为快速鉴定和使用针对SpCas9和下一代CRISPR相关核酸酶的小分子抑制剂奠定了基础。靶向CRISPR相关核酸酶的小分子抑制剂具有广泛应用于基础研究、生物医学和国防研究以及生物技术应用的潜力。”
当前,SpCas9正在开发作为多种疾病(包括艾滋病、视力障碍、肌肉萎缩症和其他遗传性疾病)的基因治疗试剂。但是,这些治疗应用将极大地受益于对SpCas9活性的剂量和时间安排进行精确控制以减少脱靶效应。控制SpCas9活性的这些方面也可能使其他应用受益,比如对模型生物的DNA进行高效编辑来构建疾病模型和研究疾病,以及在基因工程蚊子中使用基因驱动来遏制疟疾和其他蚊子传播疾病。
对SpCas9的剂量和时间控制的需求已产生了对抗CRISPR分子的需求。尽管存在靶向SpCas9的抗CRISPR蛋白,但是它们是大分子,不易渗透到细胞中,起着不可逆的作用,可被蛋白酶分解,并且可能在体内存在引起不良免疫反应的风险。相反,小分子抑制剂在蛋白水解上是稳定的,可逆的,通常是非免疫原性的,并且能够通过被动扩散轻松地递送到细胞中。此外,它们可以低成本地大规模合成,具有很小的批间差异。
在这项新的研究中,Choudhary及其团队推出了一个强大,灵敏且可扩展的平台,用于快速、经济地鉴定和验证SpCas9的小分子抑制剂。鉴于SpCas9酶的特性,以高通量方式测量CRISPR-Cas9活性从而进行药物筛选一直是具有挑战性的。为此,Choudhary团队分别开发了针对SpCas9-DNA结合和SpCas9 DNA切割活性的高通量初级和二级测定方法。对于初级测定,他们使用一种称为荧光偏振的生物化学技术来监测SpCas9与含有PAM序列的经过荧光团标记的DNA片段之间的相互作用。在二级测定中,他们使用自动显微镜来测量在细胞中由SpCas9介导的对报告基因进行DNA切割后产生的荧光变化。
通过使用这些测定方法,这些研究人员首先筛选了多种类型小分子的代表成员,以确定其成员经常抑制SpCas9的小分子类型。他们鉴定出两种先导化合物,它们以剂量依赖性方式破坏了哺乳动物细胞中SpCas9结合DNA和抑制SpCas9介导的DNA切割的能力。鉴于这些小分子阻断这种酶结合DNA,因此它们还抑制SpCas9的催化活性受到破坏的编辑技术,包括用于转录激活的那些技术,而且在人血浆中是稳定的。
Choudhary说,“这些结果为对CRISPR-Cas9活性的精确化学控制奠定了基础,从而能够安全地使用这些技术。然而,这些分子还没有为人类应用做好准备,也没有在生物体内进行功效测试。”
在未来的研究中,这些研究人员计划鉴定这些抑制剂在SpCas9:gRNA复合物上的结合位点,研究它们的作用机制,并优化它们的功效。他们还将确定这些分子是否与哺乳动物细胞中的其他靶标相互作用,并评估它们对其他的CRISPR相关核酸酶的特异性。这项新研究的早期结果表明这些分子对它们的靶标极具特异性,这是因为它们对与SpCas9的亲缘关系较远的CRISPR相关酶Cas12a没有影响。
3. Science子刊:我国科学家开发出一种可远程控制的基因编辑平台
DOI: 10.1126/sciadv.aav7199.
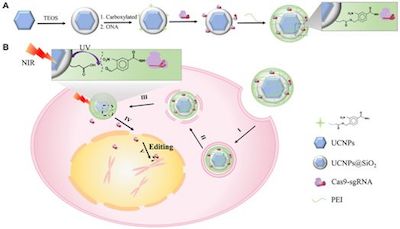
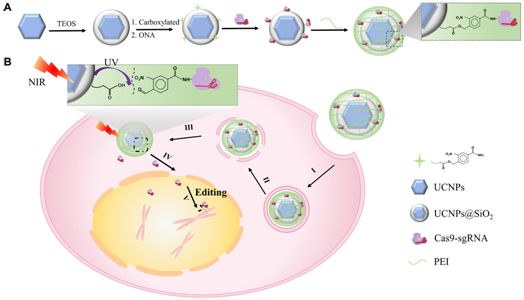
在一项新的研究中,来自中国南京大学、南京工业大学和厦门大学的研究人员开发出利用病毒将CRISPR-Cas9基因编辑工具运送到特定细胞中的一种替代方法,它涉及使用两种类型的光。相关研究结果发表在2019年4月3日的Science Advances期刊上,论文标题为“Near-infrared upconversion–activated CRISPR-Cas9 system: A remote-controlled gene editing platform”。在这篇论文中,他们描述了他们的新型载体以及它在试验用小鼠中的效果。论文通讯作者为南京大学的宋玉君(Yujun Song)教授、南京工业大学的王玉珍(Yuzhen Wang)副研究员和厦门大学的林友辉(Youhui Lin)副教授。
CRISPR-Cas9基因编辑工具是治疗遗传疾病的一场即将到来的革命,科学家们继续在各种应用中测试它的能力。鉴于当前的方法使用病毒将这种基因编辑工具递送到特定细胞中,一个研究领域涉及寻找它的替代载体系统。人们较早地就已知道这种病毒递送方法不可行,这是因为免疫系统可能会做出反应,或者更糟的是,这存在着触发肿瘤产生的风险。在这项新的研究中,中国研究人员提出了一种全新的方法:利用两种光来递送这种基因编辑工具。
他们的载体系统由对低能近红外辐射(NIR)敏感并发出紫外光的上转换纳米颗粒(upconversion nanoparticle, UCNP)组成。当近红外光照射在这些上转换纳米颗粒上时,这种光被吸收并转换成紫外光,所产生的紫外光会发射出去。
在细胞内部,这种载体系统可通过给皮肤照射近红外光加以激活。照射的近红外光穿过皮肤进入体内,并前去寻找这种载体系统。当近红外光被上转换纳米颗粒转化为紫外光时,它切割这种载体系统中的分子,从而释放出这种基因编辑工具来完成它的作用。
在实际的实验中,这些研究人员通过注射将CRISPR-Cas9工具直接递送至小鼠内部的癌性肿瘤中。当它安全就位时,他们将近红外光照射到位于肿瘤(和基因编辑工具)所在部位上方的皮肤上。当所产生的紫外光释放出这种基因编辑工具时,它开始编辑一种允许肿瘤生长的蛋白,最终结果就是肿瘤尺寸减小。
这些研究人员表示,他们的研究不仅表明一种基于光的载体能够与CRISPR-Cas9基因编辑一起发挥作用,而且还表明它能够安全地发挥作用并且提供直接的益处。
4. Nature:开发出Cas9-MMEJ可编程基因编辑方法,有望治疗143种由DNA微重复引起的疾病
DOI: 10.1038/s41586-019-1076-8
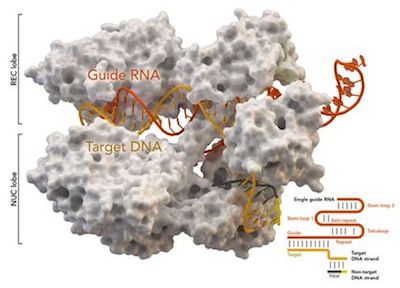
在一项新的研究中,来自美国马萨诸塞大学医学院的研究人员开发出一种利用CRISPR-Cas9和一种很少使用的DNA修复途径编辑和修复一种特定类型的与微重复(microduplication)相关的基因突变。这种可编程基因编辑方法克服了之前在基因校正中所遭遇的低效率。相关研究结果于2019年4月3日在线发表在Nature期刊上,论文标题为“Precise therapeutic gene correction by a simple nuclease-induced double-stranded break”。
论文共同通讯作者、马萨诸塞大学医学院分子、细胞与癌症生物学教授Scot A. Wolfe博士说,“这就像击中重置按钮(reset button)一样。我们不需要添加任何校正性的遗传物质,而是细胞将DNA重新拼接在一起,并移除微重复。这是基因校正的捷径,具有潜在的治疗吸引力。”
微重复是染色体发生变化而使得 DNA上的小片段被拷贝或复制。在某些基因中,当添加的核苷酸数量不能被3整除时,这些微重复就能够导致所谓的“移码突变”。这改变了基因向蛋白的翻译,从而导致功能丧失。由微重复引起的移码突变导致多达143种不同的疾病,包括肢带肌营养不良(limb-girdle muscular dystrophy)、赫曼斯基-普德拉克综合征(Hermansky-Pudlak syndrome)和家族黑蒙性白痴病(Tay-Sachs)。
Wolfe博士是CRISPR-Cas9和其他基于可编程核酸酶的基因编辑方法的专家。大多数这些技术都需要在缺陷基因处产生DNA链断裂并引入校正性的遗传物质。将新序列插入到DNA断裂中,并通过在细胞中发现的一种称为同源介导修复(homology-directed repair, HDR)的的先天性DNA修复机制进行修复。尽管在治疗上有希望,但是这种校正基因的方法是低效的并且具有其他的技术挑战。
Wolfe和论文共同通讯作者、马萨诸塞大学医学院威尔斯通肌肉营养不良中心主任、神经学教授Charles P Emerson Jr.博士认为可能存在更为直接的方法来校正由微重复引起的疾病。他们推断如果微同源介导的末端连接(microhomology-mediated end joining, MMEJ)途径可以被有效利用,而不是利用同源介导修复途径,它将会移除重复序列并恢复基因的功能序列。
与其他的细胞修复机制相比,MMEJ途径的效率更低,也更稀有。MMEJ途径通常会导致DNA断裂处的两侧发生缺失,而且MMEJ途径只负责一小部分DNA修复—据一些估计,不到10%的DNA修复。
Emerson博士有一个很有希望的疾病目标,用于评估这种编辑方法的可行性—由TCAP基因中的微重复引起的2G型肢带肌营养不良(LGMD2G)。Emerson实验室和Wolfe实验室构建的酿脓链球菌Cas9核酸酶(Strestococcus pyogenes Cas9, SpCas9)靶向TCAP基因的微重复中心附近的DNA断裂。他们接着利用SpCas9处理了源自LGMD2G患者的多能性干细胞。正如他们预测的那样,MMEJ修复机制移除了这种微重复的一个拷贝—有效地将DNA重新拼接在一起,拼接效率非常高,因而去除了突变的遗传物质并让这个基因得到恢复,从而能够产生正常的TCAP蛋白。
Emerson说,“对TCAP基因微重复进行基因编辑的简单性和高效性是一个非常激动人心的发现时刻,这就为当前无法治疗的LGMD2G开发一种治疗方法提供了一个独特的机会,这已成为我们的近期目标。”
有多少种由微重复引起的疾病可能利用MMEJ核酸酶基因编辑加以治疗呢?通过与马萨诸塞大学医学院儿科副教授Christian Mueller博士合作,这些研究人员证实与赫曼斯基-普德拉克综合征1型相关的HPS1基因中的微重复能够在患者细胞中加以校正。马萨诸塞大学医学院神经学助理教授Oliver King博士随后开发出计算方法来搜索人类基因组数据库,鉴定出143种与微重复相关的疾病可能能够利用他们的Cas9-MMEJ方法加以治疗。
Wolfe说,“从这样一个平常的开始,我们相信这种基于MMEJ的治疗策略的简单性、可靠性和有效性可能允许为许多当前无法治疗的疾病开发出基于核酸酶的基因校正疗法。”
5. Cell:操纵SKN-1A的蛋白序列编辑有望治疗阿尔茨海默病等神经退行性疾病
doi:10.1016/j.cell.2019.03.035.
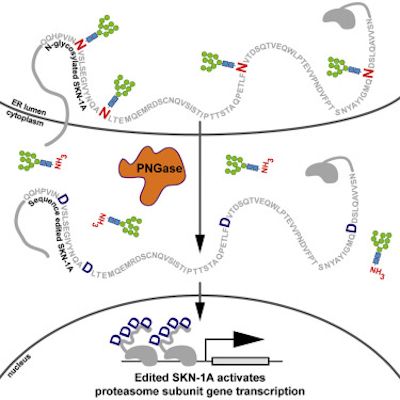
在一项新的研究中,来自美国麻省总医院的研究人员发现细胞通过编辑一种关键的传感蛋白的氨基酸序列来感知蛋白酶体功能障碍并以一种之前未描述的方式作出反应的机制。相关研究结果发表在2019年4月18日的Cell期刊上,论文标题为“Protein Sequence Editing of SKN-1A/Nrf1 by Peptide:N-Glycanase Controls Proteasome Gene Expression”。
作为一种降解不需要的或者有缺陷的蛋白的细胞组分,蛋白酶体由约20种蛋白组成,这些蛋白形成一种结构,在这种结构中,不需要的细胞蛋白以一种以受到高度调节的方式被处理掉。蛋白酶体功能障碍可导致异常蛋白的沉积,这种异常蛋白的沉积是阿尔茨海默病等神经退行性疾病的特征,也可在正常衰老中观察到。
为了应对蛋白酶体功能障碍,健康细胞增加蛋白酶体的蛋白组分的产生。两年前,麻省总医院的Gary Ruvkun博士和Nicolas Lehrbach博士已鉴定出包括转录因子SKN-1A在内的一系列传感和信号传导蛋白允许秀丽隐杆线虫中的细胞检测蛋白酶体功能障碍并对此作出反应。在这项新的研究中,Ruvkun及其团队描述了SKN-1A及其哺乳动物同源蛋白Nrf1是如何通过添加一种称为聚糖的糖分子进行修饰的,这种修饰在细胞分泌的蛋白中是比较常见的,但在DNA结合调控蛋白中是很少见的。
在正常情况下,SKN-1A/Nrf1被蛋白酶体有效降解,这就使得这种蛋白成为蛋白酶体功能的一种天然的监测者。如果蛋白的过量聚集超过蛋白酶体的处理能力,那么SKN-1A/Nrf1就不会遭受完全降解,就与蛋白酶体基因附近的DNA结合,从而诱导额外的蛋白酶体产生直到这种蛋白再次被充分降解。
2016年,Lehrbach和Ruvkun已证实SKN-1A/Nrf1的活化需要对这种蛋白进行酶促切割以及添加和随后去除聚糖分子。但是,这些事件的功能意义尚不清楚。在这项新的研究中,他们发现这种从SKN-1A中移除聚糖分子的蛋白— PNG-1—不仅移除了这些聚糖分子,而且还编辑了SKN-1A中的氨基酸序列:将4个天冬酰胺残基转化为天冬氨酸。如果将天冬酰胺转化为一种不同的氨基酸,那么SKN-1A就会出现功能障碍。在PNG-1不存在下,通过基因手段导入天冬氨酸分子也可激活SKN-1A,这表明这种蛋白序列编辑而不是去糖基化是SKN-1A发挥功能的关键。这种新发现的通过氨基酸序列编辑控制蛋白酶体功能的机制代表了内源性蛋白的一种前所未有的翻译后修饰。
这些研究人员还发现,通过基因工程手段让SKN-1A过度活化,因而在遗传上将4个去糖基化的天冬酰胺改变为天冬氨酸,这对蛋白酶体维持和抗逆性产生显著影响。过度活跃的SKN-1A赋予对蛋白酶体抑制剂的极强抵抗力,并有效地“治愈”阿尔茨海默病线虫模型。
这些研究人员指出这一发现在癌症、衰老、神经退行性疾病和一种罕见的涉及蛋白NGLY1(线虫蛋白PNG-1的人类同源蛋白,它影响着很多神经细胞的功能)突变的人类疾病中具有重要的应用。在这项新的以秀丽隐杆线虫为研究对象的研究中,他们取得的发现表明这种人类疾病还涉及未能将Nrf1中的糖分子修饰的天冬酰胺编辑成天冬氨酸,从而使得蛋白酶体未能对蛋白聚集做出反应。采取对这些天冬酰胺进行预编辑的干预措施可能重新激活受影响患者的蛋白酶体基因。
Lehrbach说,“迄今为止,我们还不明白为何SKN-1A/Nrf1的序列编辑是它调节蛋白酶体基因表达所必需的。更深入的机制理解可能有助于开发治疗NGLY1缺乏症和其他神经退行性疾病的方法。寻找受到去糖基化依赖性序列编辑调控的其他蛋白也将是令人关注的。”
6. Nature:震惊!CRISPR碱基编辑器能够诱导大量的脱靶RNA编辑
doi:10.1038/s41586-019-1161-z.
在一项新的研究中,来自美国麻省总医院、哈佛医学院和哈佛大学陈曾熙公共卫生学院的研究人员报道近期开发的几种在单个DNA碱基中产生靶向变化的碱基编辑器能够在RNA中诱导广泛的脱靶效应。他们还描述了对碱基编辑器变体进行基因改造可显著降低RNA编辑的发生率,这同时也会增加在靶DNA编辑的精确度。相关研究结果于2019年4月17日在线发表在Nature期刊上,论文标题为“Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors”。
论文通讯作者、麻省总医院病理学系的J. Keith Joung博士说道,“大多数关于脱靶基因编辑的研究都集中在DNA上,但是我们发现这种技术也可以诱导大量的RNA改变。这一令人吃惊的发现表明,当考虑碱基编辑器在细胞中的不想要的脱靶效应时,需要考虑的不仅仅是基因变化。我们还发现构建选择性地降低脱靶RNA编辑同时保留想要的在靶DNA编辑的变体来减少这些影响是可行的。”
与CRISPR-Cas基因编辑核酸酶—它诱导靶双链DNA断裂,从而导致基因变化—不同的是, CRISPR碱基编辑器能够改变DNA链中的单个核苷酸而不用诱导这种双链DNA断裂。Joung解释道,如果可以将CRISPR-Cas核酸酶比作为剪刀,那么就可将碱基编辑器比作为铅笔。当使用CRISPR-Cas修饰形式的融合蛋白靶向结合到目标位点上时,碱基编辑器使用一种称为脱氨酶的酶修饰一个特定核苷酸,从而产生可导致特定DNA改变的变化—比如,将胞嘧啶改变为胸腺嘧啶。
虽然大多数科学家都专注于碱基编辑器的DNA编辑活性,但是最常用的胞嘧啶→胸腺嘧啶编辑器(C→T编辑器)中的脱氨酶最初是因它的修饰RNA的能力而被鉴定出的。这导致Joung及其团队研究它是否可能诱导脱靶RNA效应。他们在肝脏和胚胎肾细胞系中的实验表明,虽然他们测试的这种常用的碱基编辑器在靶DNA位点上诱导高效的编辑,但是它也导致整个转录组—在细胞中发生转录的全部RNA—中发生数万个胞嘧啶→尿嘧啶(C→U)编辑。当测试一种较新的腺嘌呤靶向碱基编辑器时,他们发现了类似的结果。
为了研究减少或消除不需要的RNA编辑的可能性,Joung团队筛选了16种具有脱氨酶改造版本的碱基编辑器(即碱基编辑器改造版本),从中鉴定出两种碱基编辑器改造版本与它们的原始版本同样高效地诱导在靶DNA编辑,同时诱导显著少的RNA编辑。实际上,这些SECURE(SElective Curbing of Unwanted RNA Editing, 选择性抑制不需要的RNA编辑)变体甚至要比未经基因改造的脱氨酶更精确地诱导所需的DNA编辑。
论文第一作者Julian Grünewald博士说,“我们利用这两类碱基编辑器观察到的数以万计的RNA编辑和这些变化发生的频率感到非常吃惊。我们也很高兴看到我们能够通过使用我们的SECURE碱基编辑器变体大幅降低这些不需要的RNA编辑。”
Joung指出,研究这些RNA编辑对CRISPR碱基编辑的实验和临床应用的任何潜在影响是他的团队正在采取的重要下一步。“我们发现,我们研究的这种广泛使用的胞嘧啶碱基编辑器当在一种人细胞系中表达时对细胞活力具有适度的影响,而SECURE变体则不会。对研究应用而言,正在使用碱基编辑器的科学家们将需要在他们的实验中考虑潜在的RNA 脱靶效应。对治疗性应用而言,我们的研究结果进一步论证了将碱基编辑器表达的持续时间限制在尽可能短的时间内,以及在安全评估中考虑RNA 脱靶效应的潜在影响并使之最小化的重要性。”
Joung补充道,“正在开展的研究工作的另一个重要领域是扩大我们的努力,以尽量减少这些不需要的脱靶RNA编辑。我们当前正在尝试设计SECURE腺嘌呤碱基编辑器并探索使用其他脱氨酶的而不是我们研究的碱基编辑器中使用的脱氨酶的胞嘧啶碱基编辑器的脱靶RNA效应。我们的目标是产生一套具有最小RNA编辑活性的碱基编辑器,从而可用于研究和治疗性应用。
7. JEM:利用CRISPR/Cas9对B细胞进行基因编辑有望开发出HIV疫苗
DOI: 10.1084/jem.20190287.
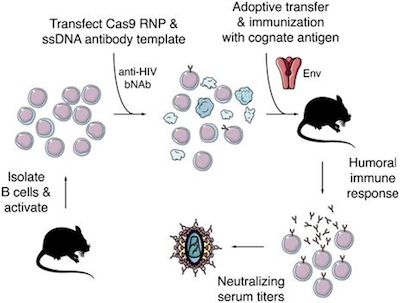
人体不能自然地保护自己免受HIV病毒感染—至少通常不能做到这一点。但是,在极少数情况下,受感染的个体会产生对抗这种病毒的广泛中和抗体(bNAb)。如今,在一项新的研究中,来自美国洛克菲勒大学的研究人员设计出一种方法,将这种对抗HIV的能力赋予给普通的免疫细胞。相关研究结果于2019年4月11日在线发表在Journal of Experimental Medicine期刊上,论文标题为“HIV-specific humoral immune responses by CRISPR/Cas9-edited B cells”。论文通讯作者为洛克菲勒大学的Michel C. Nussenzweig博士。
Nussenzweig在bNAb上的研究已产生了新的在早期临床试验中显示出前景的HIV治疗方法。如今,他着眼于第二个目标:针对这种病毒的免疫接种。
在这项新的研究中,Nussenzweig和他的同事们使用CRISPR-Cas9基因编辑技术来修饰B细胞,即一种分泌抗体的白细胞。具体而言,他们对小鼠B细胞进行基因改造,使得它们自己产生人bNAb。他们发现,以这种方式发生改变的B细胞产生的抗体水平足以保护小鼠免受HIV感染,这表明这种技术最终可能用作一种免疫工具。
虽然这项研究仍处于早期阶段,但是它证实了通过基因编辑增强免疫应答的可行性。重要的是,这种技术不会影响生殖细胞,因而避免了有时由CRISPR干预引起的伦理问题。如果能够实现,这种新的免疫方法不仅可以用于治疗HIV感染,而且还可以用于治疗任何对特定抗体敏感的疾病。
8. Blood:利用CRISPR-Cas12a基因编辑有望治疗β-地中海贫血
doi:10.1182/blood-2019-01-895094.
在一项新的研究中,来自美国达纳法伯癌症研究所、波士顿儿童医院和马萨诸塞大学医学院等研究机构的研究人员通过将CRISPR-Cas12a基因编辑应用于患者自己的造血干细胞中,开发出一种治疗一种最为常见的遗传性血液疾病—β-地中海贫血—的策略。这种方法克服了之前的技术挑战,而且要比过去更有效地对造血干细胞进行编辑。相关研究结果近期发表在Blood期刊上,论文标题为“Editing aberrant splice sites efficiently restores β-globin expression in β-thalassemia”。论文第一作者为Shuqian Xu。论文通讯作者为Daniel Bauer博士和Scot Wolfe博士。
这新的研究发现这些经过基因编辑的造血干细胞产生得到基因校正的红细胞,因而能够产生功能性的血红蛋白。
两篇论文的论文通讯作者Daniel Bauer博士说,“我们认为我们的研究确定了一种可能治愈常见的血红蛋白疾病的策略。将基因编辑与自体干细胞移植结合在一起可能是一种治疗镰状细胞病、β-地中海贫血和其他血液疾病的方法。”
根据世界卫生组织(WHO)的统计,镰状细胞病和β-地中海贫血每年在世界范围内共影响33.2万人怀孕或分娩。这两种疾病都涉及β珠蛋白编码基因发生突变。在β-地中海贫血中,突变阻止红细胞产生足够多的携氧血红蛋白分子,从而导致贫血。在镰状细胞病中,突变导致血红蛋白改变形状,使得红细胞变形为僵硬的“镰刀”形状,从而阻塞血管。
在这项新的研究中,这些研究人员使用一种类似于CRISPR-Cas9的基因编辑方案来靶向涉及剪接突变—在β-珠蛋白编码基因附近的DNA片段出现差错改变读取这个基因以组装β-珠蛋白的方式—的β-地中海贫血形式。9名β地中海贫血患者捐献了他们的造血干细胞,这样就可在培养皿中操纵它们。对于其中的一些患者,这些研究人员利用另一种不同的酶— Cas12a—来更高效地靶向这些突变。CRISPR/Cas12a高效地进行基因编辑并恢复了来自每名患者的血细胞中β-珠蛋白编码基因的正常剪接。
为临床试验做准备
Bauer认为继续追求这种方法很重要。他说,“这些疾病是非常常见的遗传性疾病,特别是在世界上资源非常有限的地区。因此,我们需要一系列广泛的治疗选择,以便为尽可能多的患者提供治疗。”
波士顿儿童医院是生物技术公司蓝鸟生物(BlueBird Bio)的股权持有者,而且一些作者申请了与治疗性基因编辑相关的专利。
如果这种处于研究中的技术经证实是有益的,那么波士顿儿童医院可能会获得经济利益。与所有研究一样,波士顿儿童医院已采取并将继续采取一切必要措施,以确保研究对象的安全性,以及本研究所获得信息的有效性和完整性。
9. Nat Med:优化的CRISPR-Cas9基因编辑有望治疗镰状细胞病
doi:10.1038/s41591-019-0401-y.
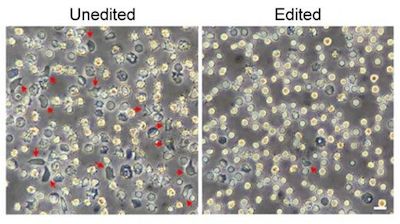
在一项新的研究中,来自美国达纳法伯癌症研究所、波士顿儿童医院和马萨诸塞大学医学院等研究机构的研究人员通过将CRISPR-Cas9基因编辑应用于患者自己的造血干细胞中,开发出一种治疗一种最为常见的遗传性血液疾病—镰状细胞病(sickle cell disease)—的策略。这种方法克服了之前的技术挑战,而且要比过去更有效地对造血干细胞进行编辑。相关研究结果于2019年3月25日在线发表在Nature Medicine期刊上,论文标题为“Highly efficient therapeutic gene editing of human hematopoietic stem cells”。论文通讯作者为Daniel Bauer博士,论文第一作者为Yuxuan Wu和Jing Zeng。
这项新的研究发现这些经过基因编辑的造血干细胞产生得到基因校正的红细胞,因而能够产生功能性的血红蛋白。
Bauer说,“我们认为我们的研究确定了一种可能治愈常见的血红蛋白疾病的策略。将基因编辑与自体干细胞移植结合在一起可能是一种治疗镰状细胞病、β-地中海贫血和其他血液疾病的方法。”
根据世界卫生组织(WHO)的统计,镰状细胞病和β-地中海贫血每年在世界范围内共影响33.2万人怀孕或分娩。这两种疾病都涉及β珠蛋白编码基因发生突变。在β-地中海贫血中,突变阻止红细胞产生足够多的携氧血红蛋白分子,从而导致贫血。在镰状细胞病中,突变导致血红蛋白改变形状,使得红细胞变形为僵硬的“镰刀”形状,从而阻塞血管。
这项新的研究使用了CRISPR-Cas9技术,特别是马萨诸塞大学医学院的Scot Wolfe博士领导的一个研究团队进行基因修饰过的Cas9蛋白,来优化基因编辑。在之前对人造血干/祖细胞的基因组进行编辑的尝试中,一旦将这些基因编辑的细胞植入骨髓中,基因编辑的效率、特异性和长期稳定性就会发生变化。这种新技术提高了基因编辑的靶向性和持久性。
Wolfe说,“对造血干细胞群体进行高效编辑—理想情况下接近100%—对在患者中实现持久的治疗效果至关重要。通过科学界多个实验室的贡献,朝着这一目标的进展一直在推进。我的研究团队与Bauer实验室合作,致力于提高CRISPR-Cas9技术的递送和进入细胞核效率,以便实现对整个造血干细胞群体的近乎彻底的治疗性编辑。”
Bauer团队使用这种策略进行高度针对性的基因编辑。之前在波士顿儿童医院的研究已表明,让一种名为BCL11A的基因失活允许红细胞即便在出生后也会继续产生胎儿形式的血红蛋白。胎儿血红蛋白不会产生镰刀形状,能够代替有缺陷的“成年”血红蛋白。最近,Bauer发现了一个更安全的靶标:BCL11A基因的增强子,仅在红细胞中有活性。
Bauer说,“通过使用我们开发的这种新型的非常有效的方法,我们能够在我们收集的几乎所有的造血干细胞中对BCL11A的增强子进行编辑,从而克服了对这些细胞进行基因编辑所面临的一些技术挑战。在我们的实验中,95%以上的增强子序列拷贝以我们期望的治疗方式发生改变。”
这种策略使得携带来自镰状细胞病患者的造血干细胞的小鼠能够产生具有足够的胎儿血红蛋白的红细胞,从而阻止红细胞产生镰刀形状。Bauer团队发现这些经过基因编辑的造血干细胞在移植到骨髓中后产生得到基因校正的红细胞。随后,当从这些小鼠中分离出造血干细胞并移植到其他小鼠中时,这些造血干细胞再次定植并且仍然携带治疗性的基因变化。
当将这一策略应用于来自β-地中海贫血患者的造血干细胞时,它恢复了构成血红蛋白的珠蛋白链的正常平衡。
为临床试验做准备
这些研究人员正在采取措施将他们开发的BCL11A增强子编辑策略应用于临床。他们正在开发一种临床级、规模化的细胞产品制造方案,并进行获得美国食品药品管理局(FDA)批准所必需的安全性研究。他们计划从美国心肺血液研究所的镰状细胞病治愈计划中寻求资金资助,以便在患者中开展临床试验。
达纳法伯癌症研究所/波士顿儿童医院癌症血液疾病中心已开始进行对镰状细胞病进行基因治疗的临床试验。该方法通过将患者的造血干细胞暴露在一种携带着指令的慢病毒中来抑制红细胞前体细胞中的BCL11A基因表达,从而增加胎儿血红蛋白的产生。
Bauer认为继续追求这种方法很重要。他说,“这些疾病是非常常见的遗传性疾病,特别是在世界上资源非常有限的地区。因此,我们需要一系列广泛的治疗选择,以便为尽可能多的患者提供治疗。”
波士顿儿童医院是生物技术公司蓝鸟生物(BlueBird Bio)的股权持有者,而且一些作者申请了与治疗性基因编辑相关的专利。
如果这种处于研究中的技术经证实是有益的,那么波士顿儿童医院可能会获得经济利益。与所有研究一样,波士顿儿童医院已采取并将继续采取一切必要措施,以确保研究对象的安全性,以及本研究所获得信息的有效性和完整性。
10. bioRxiv:成功利用CRISPR-Cas9对爬行动物进行基因编辑
doi:10.1101/591446.

在一项新的研究中,来自美国佐治亚大学的研究人员发现一种可在爬行动物身上使用CRISPR-Cas9基因编辑工具的方法。相关研究结果近期发表在bioRxiv预印版服务器上,论文标题为“CRISPR-Cas9 Gene Editing in Lizards Through Microinjection of Unfertilized Oocytes”。在这篇论文中,他们描述了他们开发的这种技术,以及它在测试的蜥蜴中的效果。
随着科学家们试图更多地了解CRISPR-Cas9本身、它的工作机制及其潜在的应用,它已被用于各种各样的实验。不过,在早期,科学家们认为CRISPR不能与爬行动物一起使用,这是因为爬行动物具有独特的生殖系统—比如,雌性蜥蜴储存精子,仅在最方便的使用加以使用,这就使得注射CRISPR-Cas9工具较为困难(如果不是不可能的话)。此外,还存在将针插入卵壳而不损坏它和阻止胚胎发育的问题。但是,存在困难并未意味着不可能,正如这些研究人员发现的那样,他们开发出一种成功的解决方法:利用CRISPR-Cas9对几只蜥蜴中的细胞进行基因编辑。
为了在蜥蜴中使用CRISPR-Cas9,这些研究人员切开了几只测试的雌性蜥蜴并在受精前将这种编辑工具直接注射到卵子中,同时这些卵子仍然存在于母体的卵巢中,然后顺其自然发育。总之,他们将这种编辑工具注射到21只蜥蜴的146个卵子中。在他们的实验中,CRISPR-Cas9经编程后对酪氨酸酶编码基因进行编辑,其中这个基因负责确定蜥蜴的颜色—当它受到失活时,蜥蜴将患上白化病(albino)。他们报道,这种编辑技术成功了四次—4只患有白化病的蜥蜴出生了。他们指出他们的技术应当也适用于其他种类的蜥蜴。
在仔细研究患上白化病的蜥蜴中实际发生的情形之后,这些研究人员发现这些蜥蜴后代具有来自父本和母本的经过编辑的基因—这意味着CRISPR-Cas9在雌性蜥蜴中保持活性的时间比预期中的要长,从而导致父本基因在受精后的变化已经发生了。(生物谷Bioon.com)

 基因君官网
基因君官网