基因编辑技术中,以锌指核酸酶(zinc-finger nucleases, ZFN)和TALEN (transcription activator-like effector nucleases)为代表的序列特异性核酸酶技术以其能够高效率地进行定点基因组编辑,在基因研究、基因治疗和遗传改良等方面展示出了巨大的潜力。
CRISPR/Cas9是继ZFN和TALEN之后出现的第三代“基因组定点编辑技术”。与前两代技术相比,其成本低、制作简便、快捷高效的优点,让它迅速风靡于世界各地的实验室,成为科研、医疗等领域的有效工具,而且经过不断改进后,更被认为能够在活细胞中最有效、最便捷地“编辑”任何基因。
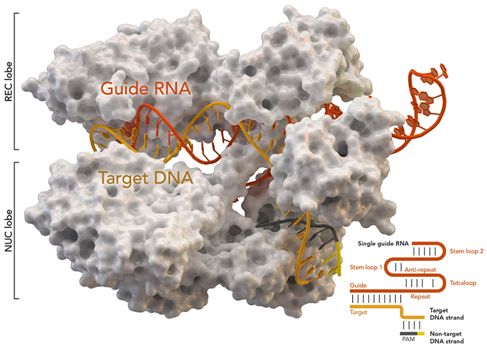
基因组编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一,受到人们的高度重视。CRISPR是规律间隔性成簇短回文重复序列的简称,Cas是CRISPR相关蛋白的简称。CRISPR/Cas9是由一种原始的细菌免疫系统改编而成的,它的作用方式是首先在基因组的一个靶位点上切割双链DNA。
在CRISPR/Cas9系统中,酶Cas9在DNA靶位点上进行切割,其中这种靶位点是这样确定的:一种被称作CRISPR RNA(crRNA)的RNA分子利用它的一部分序列与另一种被称作tracrRNA的RNA分子通过碱基配对结合在一起,形成嵌合RNA(tracrRNA/crRNA),然后,借助crRNA的另一部分序列与靶DNA位点进行碱基配对,以这种方式,这种嵌合RNA就能够引导Cas9结合到这个靶位点上并进行切割。在实际应用时,人们可以将tracrRNA和crRNA作为两种向导RNA(gRNA)或者融合在一起形成单向导RNA(single guide RNA, sgRNA),并被用来引导酶Cas9结合到靶DNA序列上并进行切割,其中Cas9与sgRNA一起被称作Cas9-sgRNA系统。
此外,CRISPR/Cas9系统靶向识别和切割与前间隔序列邻近基序(protospacer adjacent motif, PAM)相邻的特定DNA位点。作为一种最为频繁用于基因组编辑的Cas9酶,来自酿脓链球菌(Streptococcus pyogenes)的Cas9(SpCas9)仅识别作为PAM的NGG序列(简称NGG PAM,其中N代表任何一种碱基),这就限制了基因组中能够被靶向的区域。
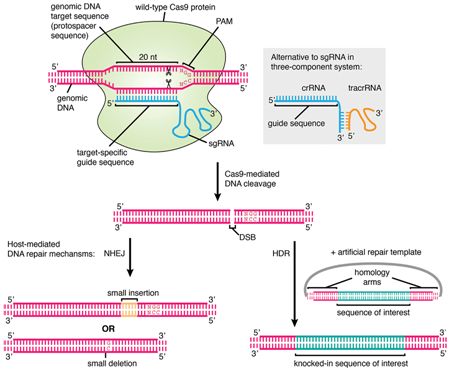
CRISPR/Cas9作用机制. 图片来自Frontiers in Genetics, 24 September 2015, doi:10.3389/fgene.2015.00300。
与CRISPR/Cas9相比,碱基编辑并不切割DNA双螺旋,而是在组成DNA或RNA的四个碱基中,利用酶精确地重新排列其中的一个碱基上的一些原子,从而将这个碱基转化为一个不同的碱基,同时不改变其周围的碱基。这种能力大大增加了改变遗传物质的选择手段。2017年,通过碱基编辑器编辑单个碱基的技术入选2017年《科学》杂志“科学十大突破”。
在2018年,科学家们在基因编辑取得重大的进展,让我们一起看看这个领域在这一年里取得的重大发现。
2018年12月,鉴于9p21.3单倍型是目前世界上已知最具影响力的心血管疾病遗传原因,Valentina Lo Sardo等人收集了来自携带着9p21.3单倍型高风险版本或低风险版本的人的血液,并让血液中的细胞经过重编程后产生诱导性多能干细胞(iPS细胞),随后利用称为转录激活因子样效应物核酸酶(TALEN)的分子剪刀对产生的iPS细胞进行基因修饰,从而移除供者细胞基因组中的9p21.3单倍型高风险或低风险版本[1]。接下来,他们诱导这些经过基因编辑的ips细胞变成血管平滑肌细胞。结果表明利用TALEN剔除9p21.3单倍型高风险版本会拯救血管平滑肌细胞的增殖、粘附和收缩。
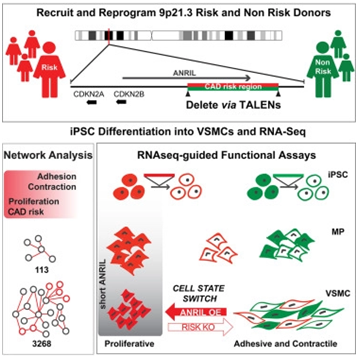
图片来自Cell, doi:10.1016/j.cell.2018.11.014。
2018年11月,Felicity Allen等人在一项迄今为止最大规模的探究CRISPR作用机制的研究中,发现对经过CRISPR-Cas9切割的DNA的修复依赖于靶DNA和gRNA的精确序列,并且发现在相同的序列中,细胞的这种修复机制是可重复的[2]。他们随后利用大量的序列数据开发出一种称为FORECasT的机器学习计算工具,该工具能够仅使用靶DNA序列来预测修复后的DNA序列。与此同时,HMax W. Shen等人观察细胞如何修复小鼠和人类基因组中CRISPR靶向切割的2000个位点,并将所获得的数据输入到一种称为inDelphi的机器学习模型中,从而促进这种算法学习细胞如何对每个位点上的切割作出反应,它的预测结果表明在很多位点上,经过校正的基因并不包含大量的变异,而是一种单一的结果,如校正致病性的基因[3]。
2018年11月,Haibao Zhu等人将磁性纳米颗粒(MNP)与重组的圆柱形杆状病毒载体(baculovirus vector, BV)相结合,开发出运送CRISPR/Cas9的MNP-BV载体,选择杆状病毒载体是因为是它具有较大的装载能力,而且在局部磁场的作用下能够克服补体系统导致的杆状病毒载体局部失活[4]。施加局部的磁场能够高效地靶向运送MNP-BV载体,并促进将MNP-BV载体转导到细胞中,从而实现通过空间控制对特定组织或器官中的基因进行修饰。
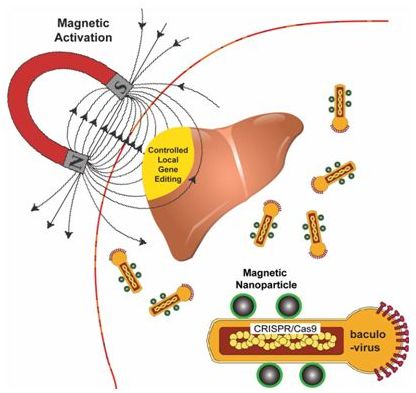
图片来自Nature Biomedical Engineering, doi:10.1038/s41551-018-0318-7。
2018年10月,Lucas B. Harrington等人发现了迄今为止最小的CRISPR基因编辑系统:在从科罗拉多州来复镇(Rifle)的一个有毒的净化场所获得的地下水样品中经过测序的古细菌基因组中发现了Cas14蛋白[5]。与Cas9一样,Cas14具有作为生物技术工具的潜力。由于具有较小的体积,Cas14可能用于编辑小细胞或某些病毒中的基因。不过鉴于Cas14的单链DNA切割活性,它更有可能改善目前正在开发的用于快速诊断传染病、基因突变和癌症的CRISPR诊断系统。
2018年10月,Florian Schmidt等人利用肠道细菌大肠杆菌开展研究,将来自一种不同的细菌物种的编码CRISPR-Cas9系统的基因导入到大肠杆菌中,其中的Cas9基因与一种逆转录酶融合在一起[6]。导入这些编码CRISPR-Cas9的外源基因的大肠杆菌细胞能够产生一种结合短mRNA分子的蛋白复合物。这种逆转录酶将mRNA翻译为含有与初始的mRNA相同的遗传信息的DNA,然后将它们作为间隔序列存储在CRISPR阵列中。这种过程能够多次发生,从而使得新的间隔序列以相反的时间顺序添加到CRISPR阵列,因此最近获得的DNA片段总是位于最前面,由此,就开发出开发出一种存储转录事件的细胞记录设备。在此之前,Weixin Tang等人利用CRISPR构建出一种称为CAMERA(CRISPR-mediated analog multi-event recording apparatus)的细胞事件记录技术[7]。
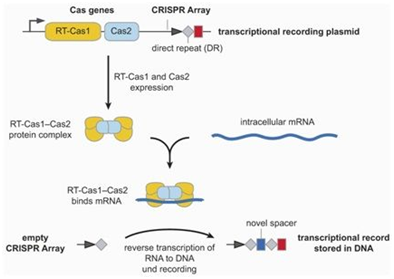
图片来自Nature, doi:10.1038/s41586-018-0569-1。
2018年9月,鉴于在临床中使用CRISPR/Cas9基因编辑的一个障碍是Cas9核酸酶可能会在错误的位点上切割DNA,Pinar Akcakaya等人开发出一种让脱靶效应最小化的方法:将基因组DNA切割为大约长300个碱基对的片段,给这些片段连接上一系列让DNA环化的接头(adapter),随后引入Cas9和gRNA的复合物,这种复合物在某些位点上切割环状DNA,从而让它线性化[8]。另一批核酸酶会降解剩余的未被切割的环状DNA。通过这种方式,这些研究人员能够对线性化的DNA进行测序,从而能够观察Cas9切割(不论是有意的还是无意的)的位点并预测gRNA是否会导致体内脱靶效应。随后,他们在小鼠中测试了他们的预测结果:当使用在体外发现的会在基因组中数千个错误位点进行切割的gRNA时,在检测的一部分的预测位点中,超过40%的位点也在小鼠肝脏中发生突变。一个位点在体外筛选中出现的频率越高,它在体内发生突变的可能性就越大。换句话说,在体外发生差错的gRNA在体内也会发生差错。
2018年9月,美国迈阿密大学的Carlos Moraes团队将含有靶向线粒体的TALEN(mitochondrial-targeted TALEN, MitoTALEN)的腺相关病毒(AAV)注射到携带着mtDNA突变的小鼠肌肉中,在六个月后,小鼠肌肉组织中的突变mtDNA的水平下降了50%以上—低于通常与线粒体疾病的症状相关的水平[9]。与此同时,英国剑桥大学的Michal Minczuk团队,研究人员将含有靶向线粒体的ZFN(mitochondrially targeted zinc-finger nuclease, mtZFN)的AAV病毒注射到小鼠的尾静脉中,从而被系统性地运送到心脏中。仅两个多月后,突变mtDNA的水平在心脏组织中下降了大约40%[10]。
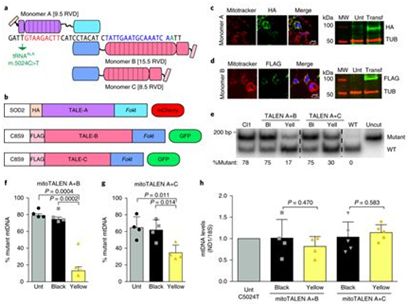
图片来自Nature Medicine, doi:10.1038/s41591-018-0166-8。
2018年9月,Hiroshi Nishimasu等人构建出一种合理设计的SpCas9变异体(SpCas9-NG),它能够识别作为PAM的NG而不是NGG[11]。这种SpCas9-NG变异体增加了基因组中的靶向范围,但是具有与野生型SpCas9类似的特异性:在人细胞中,这种SpCas9-NG变异体在携带着NG PAM的内源性靶位点中诱导碱基插入或删除(insertion or deletion, indel);将这种SpCas9-NG变异体与活化诱导的胞苷脱氨酶(activation-induced cytidine deaminase, AID)融合在一起能够调节人细胞中携带着NG PAM的靶位点上的C→T转化,即由碱基胞嘧啶(C)转化为碱基胸腺嘧啶(T)。
2018年9月,Avery C. Rossidis等人将名为BH3的碱基编辑器和CRISPR系统一起导入患有遗传性酪氨酸血症1型(HT1)的小鼠胚胎中,结果表明,接受BH3治疗的小鼠胚胎,在出生后的3个月里肝脏中经过编辑的肝细胞数量稳定[12]。在HT1小鼠模型中,BH3治疗提高了它们的肝脏功能和生存率。与此同时,Lukas Villiger等人将CRISPR/Cas9系统和胞苷脱氨酶结合在一起,将导致苯丙酮尿症的DNA碱基对C-G转变为在正常人中出现的T-A,而且接受这种碱基编辑治疗的小鼠肝脏中60%的致病基因突变能够被成功校正,从而使得小鼠体内的苯丙氨酸水平降低到正常水准,而且不再显示出任何苯丙酮尿症的症状[13]。
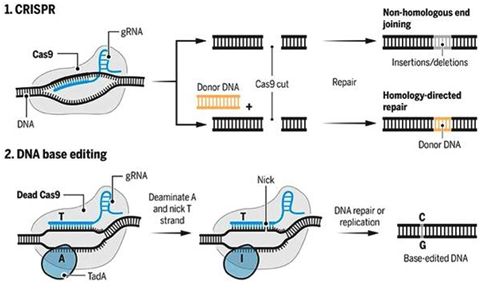
CRISPR/Cas9系统和碱基编辑器作用机制示意图,图片来自Science期刊。
2018年9月,鉴于III型CRISPR/Cas效应复合物在结合到靶RNA时会合成由4或6个腺苷一磷酸(AMP)分子连接在一起而形成的环寡腺苷酸,所产生的环寡腺苷酸诱导细胞进入抗病毒状态,但是如果长时间处于激活状态的话,细胞也会死亡,Januka S. Athukoralage等人鉴定出一种称为环状核酸酶(ring nuclease)的关闭开关,它特异性地切割这些环寡腺苷酸分子,并且当入侵的病毒遭受破坏时,它让细胞恢复到未受感染的状态[14]。环状核酸酶是CRISPR基因组工程工具包中的一个重要的新组分。这将引发遗传病和感染治疗变革。
2018年9月,Kyle E. Watters等人利用一种全面的生物学信息学和实验筛选方法鉴定出三种阻断或减少在人细胞中进行CRISPR/Cas12a介导的基因组编辑的抑制剂[15]。与此同时,Nicole D. Marino等人发现了12个Acr基因,这些基因编码的Acr蛋白包括抑制V-A型CRISPR/Cas系统和I-C型CRISPR/Cas系统的蛋白,如AcrVA1。值得注意的是,当在人细胞中进行测试时,AcrVA1最为有效地抑制Cas12a的一系列直向同源物,包括MbCas12a、Mb3Cas12a、AsCas12a和LbCas12a[16]。这些CRISPR/Cas12a抑制剂提供了对CRISPR基因编辑进行控制的生物技术工具。
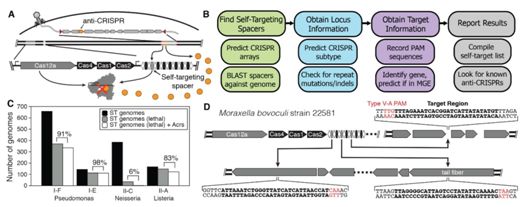
通过生物信息手段发现抑制CRISPR/Cas12a的Acr蛋白。图片来自Science, doi:10.1126/science.aau5138。
2018年8月,我国科学家Weiqi Zhang等人在全球首次实现了SIRT6在非人灵长类动物中的全身敲除,获得了世界上首例特定长寿基因敲除的食蟹猴模型[17]。与SIRT6敲除小鼠表现的加速衰老表型明显不同,SIRT6敲除的食蟹猴在出生数小时内即死亡。多项分析结果显示,SIRT6敲除的食蟹猴未见加速衰老表型,却表现出严重的全身发育迟缓。新生SIRT6敲除猴的脑及多种其他器官组织均表现出明显的胚胎期未成熟的细胞和分子特征。在此之前,我国科学家Sen Yan等人利用CRISPR/Cas9基因编辑技术将人体中导致亨廷顿舞蹈病(Huntington’s disease, HD)的具有非常长的谷氨酰胺重复序列的mHTT编码基因的一个片段导入到猪成纤维细胞中[18]。随后,体细胞核移植产生携带这种基因改变的猪胚胎,由此构建出基因敲入HD猪模型。
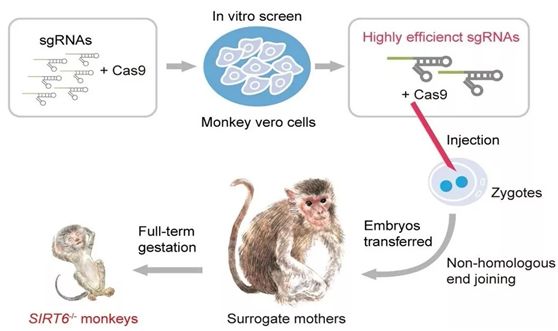
利用CRISPR/Cas9基因编辑技术获得SIRT6全身敲除的食蟹猴,图片来自Nature, doi: 10.1038/s41586-018-0437-z.
2018年7月,Alex Marson团队开发出一种强大的分子“剪切和粘贴”系统,用于重写人T细胞中的基因组序列:当某些数量的T细胞、DNA和CRISPR“剪刀”混合在一起然后暴露在一种适当的电场中时,这些T细胞将摄入DNA和CRISPR剪刀,并且精确地将特定的基因序列整合到CRISPR在基因组中的靶切割位点上[19]。它在未来有助于加速开发出新的更加安全的治疗癌症、自身免疫疾病和其他疾病(包括罕见的遗传性疾病)的疗法。
2018年7月,Michael Kosicki等人发现CRISPR/Cas9基因编辑工具能够导致基因组上的靶位点附近发生大片段DNA缺失和重排。这些重排导致之前相隔遥远的DNA序列被拼接在一起。这种现象在他们测试的所有三种细胞类型—小鼠胚胎干细胞、小鼠造血祖细胞和一种人分化细胞系—中都很普遍[20]。这些变化能够干扰对实验结果的解释,并且可能使得设计基于CRISPR的疗法的努力复杂化。
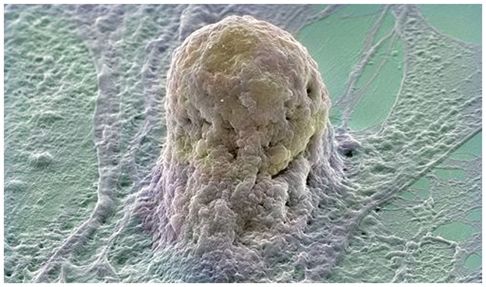
针对人胚胎
干细胞的基因编辑实验揭示出CRISPR-Cas9系统的不精确性。图片来自Annie Cavanagh via Wellcome/CC BY NC。
2018年7月,Lili Wang等人对归巢核酸内切酶(meganuclease)进行基因形式,使得它能够特异性地识别并且失活PCSK9基因,随后利用腺病毒载体(AAV)携带经过基因修饰的归巢核酸内切酶来干扰灵长类动物肝脏中的PCSK9基因,结果发现在利用中等和高剂量AAV载体治疗的动物机体中,PCSK9的水平下降了45%-84%,而且有害胆固醇的水平也下降了30%-60%[21]。
2018年7月,Demosthenes P. Morales等人开发出光触发的基因组编辑方法,这种方法的关键是空心的金纳米球,在这些金纳米球上包被着DNA报告链(发出红色荧光)和由Cre重组酶与细胞穿透肽组成的融合蛋白[22]。一旦被摄入到细胞中,这些金纳米球被包埋在内体(endosome)中。超快脉冲近红外激光 —对细胞无害且高效地穿过组织—随后照射这些被包埋的金纳米球和它们的蛋白涂层。这种照射导致这些金纳米球受到激发,这会导致纳米气泡形成,从而让内体出现开口并允许它的蛋白内含物逃出。这些蛋白如今自由地前往存储着遗传物质的细胞核中,并让细胞穿透肽进入。Cre能够寻找、剪切细胞中的双螺旋DNA,并且将DNA报告链粘贴到双螺旋DNA中。
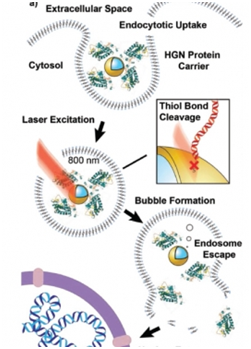
图片来自Small, doi:10.1002/smll.201800543。
2018年6月,Emma Haapaniemi等人发现在实验室环境中对人类细胞进行CRISPR-Cas9基因编辑操作或许会激活名为p53的癌症抑制蛋白,一旦被激活后,p53就会降低CRISPR-Cas9基因编辑的效率,而不携带p53或无法激活p53表达的细胞就会表现出较好的基因编辑效果,但不幸的是,缺少p53常备认为会使得细胞失控生长并且发生癌变[23]。通过挑选已经成功修复损伤基因的细胞,这可能就会在无意中选择不携带功能性p53的细胞,如果将这种细胞转移到患者体内,以此作为基因疗法来治疗遗传性疾病,这样的细胞或许就会诱发癌症。
2018年5月,Matthew G. Costales等人开发出一种经设计后能够精确和有选择性地结合特定RNA的称为RIBOTAC(ribonuclease-targeting chimeras)的技术,这种RIBOTAC复合物的第一部分是RNA降解酶RNase L,它的另一个部分是药物类似分子Targaprimir-96,用于与一种已知促进癌细胞增殖的microRNA致癌基因(即miRNA-96)结合[24]。这种RIBOTAC复合物局部激活内源性的RNase L,从而切割癌细胞中的miRNA-96前体,这又会增加促凋亡转录因子FOXO1表达,从而选择性地触发恶性肿瘤细胞死亡。
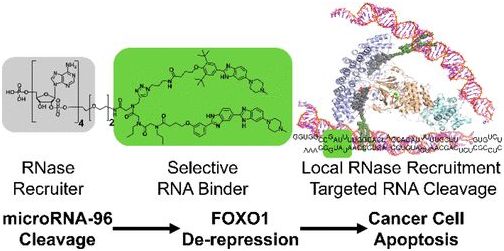
图片来自Journal of the American Chemical Society, doi:10.1021/jacs.8b01233。
2018年4月,Cameron Myhrvold等人开发出一种更加简单的方法,从而允许Cas13直接在唾液或血液等体液样品中检测它的靶标。这种方法被称作HUDSON(Heating Unextracted Diagnostic Samples to Obliterate Nucleases),通过对临床样品进行快速的化学和热处理来灭活某些会降解基因靶标的酶。这些经过处理的临床样品随后通过SHERLOCK诊断平台进行检测,最终的检测结果(阳性或阴性)能够在试纸条上很容易地观察到[25]。Feng Zhang团队对SHERLOCK诊断平台进行一系列优化,让这种平台使用来自不同细菌种类的Cas13和Cas12a(以前称为Cpf1)酶来产生额外的信号;此外,还添加了一种额外的CRISPR相关酶(即Csm6)来放大检测信号,从而增加了SHERLOCK的灵敏度,并增加准确地定量确定样品中的靶分子水平和一次测试多种靶分子的能力[26]。Jennifer A. Doudna团队观察到Cas12a锁定它的靶标并进行切割,它就会开始撕碎它能够发现的所有单链DNA,开发出一种简单的被称作DETECTR(DNA Endonuclease Targeted CRISPR Trans Reporter)的新方法,它能够实现灵敏而又准确的DNA检测[27]。这种SHERLOCK诊断平台是Feng Zhang团队在一年之前基于Cas13酶和RNA报告分子开发出来的:在经过两个扩增步骤后,当Cas13a检测到靶RNA序列时,它的无区分的RNA酶活性(即附带切割活性)也会切割这种RNA报告分子,从而释放可检测到的荧光信号[28]。

图片来自Zhang lab, Broad Institute。
2018年3月,Silvana Konermann等人构建出一种靶向RNA而不是靶向DNA的新工具,并利用它校正来自一名痴呆症患者的细胞中的蛋白不平衡,从而让它们恢复到健康水平。这种被称作CasRx的新工具来自黄化瘤胃球菌(Ruminococcus flavefaciens)XPD3002,它为科学家们提供一种强大的方法来开发新的基因疗法和研究基础的生物学功能[29]。
2018年2月,鉴于spCas9对靶DNA序列的识别依赖于特定的PAM序列—NGG序列,仅1/16的人类基因组区域含有这种PAM序列,这无疑限制了基因编辑的范围,为此,Johnny H. Hu等人开发出称为xCas9的SpCas9变体,该变体广泛识别多种PAM序列,包括NG、GAA和GAT,编辑范围至少增加了4倍,可以靶向编辑基因组的1/4区域[30]。更重要的是,相比于spCas9,xCas9错误编辑的概率降低了。
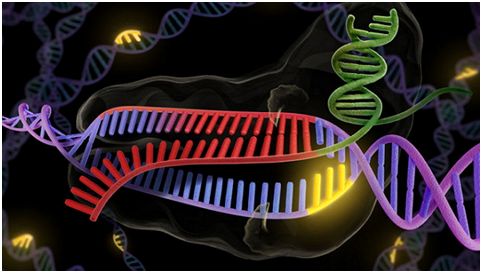
在gRNA(绿色和红色)的帮助下,CRISPR/Cas9通过结合到PAM序列(黄色)上切割靶DNA序列,图片来自KC Roeyer/University of California, Berkeley。
2018年2月,X. Shawn Liu等人鉴于脆性X染色体综合征是由患者X染色体上的FMR1基因发生突变引起,X. Shawn Liu等人开发一种移除甲基化的改进型CRISPR / Cas9系统:将没有切割活性的dCas9与甲基胞嘧啶双加氧酶Tet1融合在一起,移除FMR1基因中的三核苷酸(CGG)重复序列上的甲基化标签[31]。移除这些甲基化标签会让FMR1基因的表达恢复到正常的水平。
2018年1月,Carsten Trevor Charlesworth等人分析了22名婴儿和12名健康成年人的血液样品以便确定这些人是否对这两种最常用的Cas9酶版本—来自金黄色葡萄球菌(Staphlococcus aureus)的Cas9酶(即SaCas9)和来自酿脓链球菌(Streptococcus pyogenes)的Cas9酶(SpCas9)—产生免疫反应,结果发现79%的研究参与者对SaCas9产生抗体, 65%的研究参与者对SpCas9产生抗体[32]。此外,Sojung Kim等人也发现体外转录的gRNA(in vitro transcribed gRNA, IVT gRNA)含有5’ 三磷酸(5’ppp)基团,这会触发人细胞中的I型干扰素介导的免疫反应,以及干扰素激活的抗病毒效应蛋白(如DDX58)的表达增加,从而导致细胞死亡[33]。
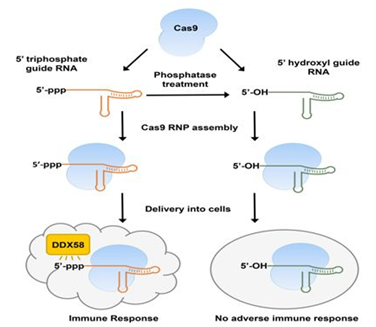
图片来自Genome Research, doi:10.1101/gr.231936.117。
其实,科学家们针对基因编辑领域的研究不胜枚举,以上罗列的仅是其中的一小部分。当然,迄今为止,涉及基因编辑的疾病治疗研究基本上局限在细胞模型和动物模型上,这是因为诸如CRISPR/Cas9之类的基因编辑技术存在着编辑效率较低和脱靶效应等缺点,因此贸然进行开展人体临床试验,会引发难以预测的结果,这不奇怪当中国科学家贺建奎声称世界上首批经过基因编辑的婴儿出生时,国内外的口诛笔伐纷至杳来。不过,在未来,科学家们将从病理学、分子生物学、基因、蛋白和组学等不同角度深入探究ZFN、TALEN和不同类型的CRISPR/Cas系统及其改进版本等基因编辑技术的详细作用机制,人们最终有朝一日能够利用基因编辑技术治疗HIV感染、癌症、血液系统疾病、神经系统疾病和遗传疾病等一系列疾病。(生物谷 Bioon.com)
参考文献:
1. Valentina Lo Sardo et al. Unveiling the Role of the Most Impactful Cardiovascular Risk Locus through Haplotype Editing. Cell, Published online: December 6, 2018, doi:10.1016/j.cell.2018.11.014.
2. Felicity Allen et al, Predicting the mutations generated by repair of Cas9-induced double-strand breaks, Nature Biotechnology (2018). DOI: 10.1038/nbt.4317.
3. HMax W. Shen, Mandana Arbab, Jonathan Y. Hsu et al. Predictable and precise template-free CRISPR editing of pathogenic variants. Nature, Published Online: 07 November 2018, doi:10.1038/s41586-018-0686-x.
4. Haibao Zhu et al. Spatial control of in vivo CRISPR–Cas9 genome editing via nanomagnets. Nature Biomedical Engineering, Published Online: 12 November 2018, doi:10.1038/s41551-018-0318-7.
5. Lucas B. Harrington et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science, Published Online: 18 October 2018, doi:10.1126/science.aav4294.
6. Florian Schmidt et al. Transcriptional recording by CRISPR spacer acquisition from RNA. Nature, Published Online: 03 October 2018, doi:10.1038/s41586-018-0569-1.
7. Weixin Tang et al. Rewritable multi-event analog recording in bacterial and mammalian cells. Science, Published online: 15 Feb 2018, doi:10.1126/science.aap8992.
8. Pinar Akcakaya et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature, Published Online: 12 September 2018, doi:10.1038/s41586-018-0500-9.
9. Sandra R. Bacman, Johanna H. K. Kauppila, Claudia V. Pereira et al. MitoTALEN reduces mutant mtDNA load and restores tRNAAla levels in a mouse model of heteroplasmic mtDNA mutation. Nature Medicine, Published Online: 24 September 2018, doi:10.1038/s41591-018-0166-8.
10. Payam A. Gammage, Carlo Viscomi, Marie-Lune Simard et al. Genome editing in mitochondria corrects a pathogenic mtDNA mutation in vivo. Nature Medicine, Published Online: 24 September 2018, doi:10.1038/s41591-018-0165-9.
11. Hiroshi Nishimasu et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 21 Sep 2018, 361(6408):1259-1262, doi:10.1126/science.aas9129.
12. Avery C. Rossidis et al. In utero CRISPR-mediated therapeutic editing of metabolic genes. Nature Medicine, October 2018, 24(10):1513–1518, doi:10.1038/s41591-018-0184-6.
13. Lukas Villiger et al. Treatment of a metabolic liver disease by in vivo genome base editing in adult mice. Nature Medicine, October 2018, 24(10):1519–1525, doi:10.1038/s41591-018-0209-1.
14. Januka S. Athukoralage et al. Ring nucleases deactivate type III CRISPR ribonucleases by degrading cyclic oligoadenylate. Nature, Published Online: 19 September 2018, doi:10.1038/s41586-018-0557-5.
15. Kyle E. Watters et al. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science, Published Online: 06 Sep 2018, doi:10.1126/science.aau5138.
16. Nicole D. Marino et al. Discovery of widespread Type I and Type V CRISPR-Cas inhibitors. Science, Published Online: 06 Sep 2018, doi:10.1126/science.aau5174.
17. Weiqi Zhang et al. SIRT6 deficiency results in developmental retardation in cynomolgus monkeys. Nature, 560:661–665 (2018) , doi:10.1038/s41586-018-0437-z.
18. Sen Yan et al. A Huntingtin Knockin Pig Model Recapitulates Features of Selective Neurodegeneration in Huntington’s Disease. Cell, Published online: March 29, 2018, doi:10.1016/j.cell.2018.03.005.
19. Theodore L. Roth et al. Reprogramming human T cell function and specificity with non-viral genome targeting. Nature, Published online: 11 July 2018, doi:10.1038/s41586-018-0326-5.
20. Michael Kosicki et al. Repair of double-strand breaks induced by CRISPR–Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology, Published online: 16 July 2018, doi:10.1038/nbt.4192.
21. Lili Wang, Jeff Smith, Camilo Breton, et al. Meganuclease targeting of PCSK9 in macaque liver leads to stable reduction in serum cholesterol. Nature Biotechnology (2018). DOI:10.1038/nbt.4182.
22. Demosthenes P. Morales et al. Light‐Triggered Genome Editing: Cre Recombinase Mediated Gene Editing with Near‐Infrared Light. Small, Published Online: 02 July 2018, doi:10.1002/smll.201800543.
23. Emma Haapaniemi et al. CRISPR–Cas9 genome editing induces a p53-mediated DNA damage response. Nature Medicine (2018). DOI: 10.1038/s41591-018-0049-z.
24. Matthew G. Costales et al. Small Molecule Targeted Recruitment of a Nuclease to RNA. Journal of the American Chemical Society, Published online:May 24, 2018, doi:10.1021/jacs.8b01233.
25. Cameron Myhrvold et al. Field-deployable viral diagnostics using CRISPR-Cas13. Science, 27 Apr 2018, 360(6387):444-448, doi:10.1126/science.aas8836.
26. Jonathan S. Gootenberg et al. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6. Science, 27 Apr 2018, 360(6387):439-444, doi:10.1126/science.aaq0179.
27. Janice S. Chen et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science, 27 Apr 2018, 360(6387):436-439, doi:10.1126/science.aar6245.
28.Jonathan S. Gootenberg et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science, 13 Apr 2017, doi:10.1126/science.aam9321.
29. Silvana Konermann et al. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell, Published online: March 15, 2018, doi:10.1016/j.cell.2018.02.033.
30. Johnny H. Hu, Shannon M. Miller, Maarten H. Geurts et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, Published online: 28 February 2018, doi:10.1038/nature26155.
31. X. Shawn Liu et al. Rescue of Fragile X Syndrome Neurons by DNA Methylation Editing of the FMR1 Gene. Cell, Published online: February 15, 2018, doi:10.1016/j.cell.2018.01.012.
32. Carsten Trevor Charlesworth et al. Identification of Pre-Existing Adaptive Immunity to Cas9 Proteins in Humans. bioRxiv, doi:10.1101/243345.
33. Sojung Kim, Taeyoung Koo, Hyeon-Gun Jee et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research, 2018, 28(3), doi:10.1101/gr.231936.117.

 基因君官网
基因君官网