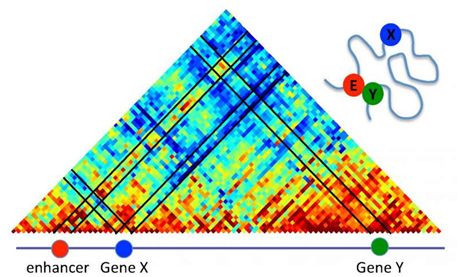
2017年3月10日/生物谷BIOON/—细胞面临着艰巨的任务。它们不得不熟练地将几米长的遗传物质塞进仅长5微米的细胞核中。这种折纸术让基因和它们的调节区域发生空间相互作用,从而能够影响人类健康和疾病。如今,一个国家研究小组开发出一种强大的新技术来绘制整个基因组的三维图谱。相关研究结果于2017年3月8日在线发表在Nature期刊上,论文标题为“Complex multi-enhancer contacts captured by genome architecture mapping”。
基因经激活后产生RNA和蛋白,然后当这些分子不再需要时,就会再次被关闭。基因和它的调节区域都是DNA序列,它们可能在线性的基因组中相隔较远的距离。这就给细胞带来挑战,这是因为这些区域通常需要接触才能够激活基因。
它也给试图理解生物学上的一个重要问题的科学家们带来困难:细胞如何决定哪些基因应当被激活,何时被激活?这种答案部分上依赖于将每个基因与它的调节序列进行匹配。但是DNA链太薄而不能够在显微镜下追踪它们,而且即便能够开展这种追踪,人们也需应付细胞核中的大量DNA。
如今,一种被称作基因组结构绘制(Genome Architecture Mapping, GAM)的新技术有助鉴定这些接触。它涉及对细胞或组织进行瞬间冻结,然后切割单个细胞核的薄片。接着,这个研究小组对每个细胞核薄片内的微量DNA进行测序,并利用一种被称作SLICE的数学模型鉴定出DNA链之间相互作用增加的“热点”。这个数学模型研究了不同的基因组区域(如基因和增强子等)出现在细胞核薄片中的频率,从而推断关于基因和激活它们的增强子的相对位置的信息。
论文共同通信作者Ana Pombo解释道,“一种类比可能是这样的;如果你想要理解学龄儿童如何与你进行互动,那么你可能在他们坐在食堂里或一起出现在操场时随机拍摄图片。如果你一个多月内拍摄多次,那么你将开始观察哪些学龄儿童经常挨着坐,哪些学龄儿童在玩耍时一起到处跑。这些随机图片可能让你知道这些学龄儿童的社交行为。”Pombo在英国医学研究委员会伦敦医学研究所开始这项研究,如今在德国马克斯-德尔布吕克分子医学中心柏林医学系统生物学研究所任职。
论文共同通信作者、意大利那不勒斯费德里克二世大学的Mario Nicodemi说,“利用数学方法从真实的相互作用中过滤出随机相遇是有可能做到的。” Nicodemi构想出这种数学模型,并且在他的博士生Antonio Scialdone的帮助下,开发出它。
在这项新的研究中,这个研究团队将这种方法用于小鼠胚胎干细胞中。他们希望它将有助揭示哪些基因的活性在一些非常严重的疾病中受到破坏。在一些疾病中,问题出现在基因序列中,但是在基因组其他地方发现的调节区域中存在的缺陷同样是危险的,而且是更难理解的。这些新的数据提供一系列新的猜测供研究人员详细检查。
尽管之前的研究已鉴定出双向接触,但是这种信息并没有揭示出这些接触多长时间发生一次,以及它们可能发生多大重要的作用。
论文共同第一作者Robert Beagrie说,“长期以来,人们一直在测量双向接触。这些研究经常表明你能够有一组不同的DNA元件,而且这些DNA元件彼此间成对地发生相互作用。利用这种新的方法,我们能够产生所有的我们自信发生群体相互作用的基因组区域的全基因组目录。”如今,这些研究人员能够可靠地定量检测发生活跃表达的基因组区域中的“三向接触(three-way contacts)”。
不过,利用GAM获得的最为显著的进展在于这些实验基于单个细胞(不论在组织中是常见的还是罕见的)开展的,并且追踪它们彼此在组织内的相对位置。现存的方法需要大量相同类型的细胞,这就使得很难研究罕见细胞类型的生物学特征和罕见的疾病类型。Pombo说,“将这种方法用于人组织样品中有巨大潜力来记录调节区域和它们的靶基因之间的接触,并且利用它理解基因变异和这种变异如何可能在某些方面改变核生物学。”
一些研究人员开始有兴趣利用这种技术探究当逆转录病毒将它们的DNA插入到宿主基因组中会发生什么。癌症科学家也热衷于构建肿瘤特定区域的DNA图谱。Nicodemi解释道,“通过探究GAM数据的独特性质,数学模型能够可靠地获得这些信息,从而为鉴定出多种可能在基因调节中发挥着关键性作用的群体相互作用。我们如今能够询问一个基因是同时接触它的所有增强子,还是一次接触一个增强子。我们知道很多在早期发育中发挥着重要作用的基因具有多种增强子。但是它们如何和为何调节基因仍然是尚待解答的问题。”(生物谷 Bioon.com)
本文系生物谷原创编译整理,欢迎转载!点击 获取授权 。更多资讯请下载生物谷APP。
原始出处:
Robert A. Beagrie, Antonio Scialdone, Markus Schueler et al. Complex multi-enhancer contacts captured by genome architecture mapping. Nature, Published online 08 March 2017, doi:10.1038/nature21411.
 基因君官网
基因君官网