2017年3月28日/生物谷BIOON/—确定细胞中的基因如何影响它的功能是分子遗传学研究的首要目标。但是大多数基因型-表型筛选受限于一项实验实际能够测量的基因扰动(genetic perturbation)数量。简言之,研究的基因扰动越多,实验就变得更加成本高昂和更加耗时。
确实,美国加州大学圣地亚哥分校的Trey Ideker说,很少开展过大规模的基因型-表型筛选,而且那些开展过的大规模筛选是庞大的工作。如今,多亏两种高度类似的技术:一种技术是由美国布罗德研究所的Aviv Regev和同事们开发出的,该技术被称作Perturb-Seq[1];另一种技术是由以色列魏兹曼研究所的Ido Amit和同事们开发出的,该技术被称作CRISP-Seq[2]。在一项实验中,利用Perturb-Seq、CRISP-Seq或将这两种技术结合在一起研究众多基因扰动是可能的[3]。
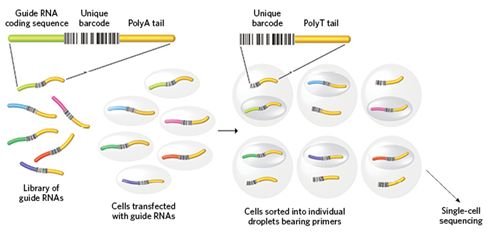
Perturb-Seq和CRISP-Seq背后的工作原理是给单个基因扰动和受到影响的细胞添加条形码以至于利用测序能够鉴定出这两者。简单地说,靶向感兴趣基因的添加独特条形码的CRISPR向导RNA文库被导入一个细胞群体中。随后利用添加独特条形码的引物将单个细胞的mRNA提取出。对这些mRNA进行测序揭示出CRISPR靶向基因和由此产生的单个细胞的转录谱。重要的是,能够对数以万计的这样的细胞进行平行测序。
Regev和Amit利用他们的技术研究了免疫细胞中的转录因子功能和分化调节,以及其他。但是,Ideker说,它们的可能应用是无穷的。他说,这是“这些技术的首个概念研究,而且它们将变得越来越好。”(生物谷 Bioon.com)
|
方法 |
基因破坏 |
转录组分析 |
估计成本* |
估计时间* |
|
基因敲除后进行转录组分析 |
可选择的基因破坏技术有很多,包括基因编辑(CRISPR)、随机突变和同源重组 |
将含有受到破坏的基因的细胞汇集在一起,提取出它们的mRNA,并进行测序 |
利用CRISPR分析1000个基因扰动需要13.5万美元 |
利用CRISPR分析1000个基因扰动需要30个小时 |
|
Perturb-Seq或CRISP-Seq |
CRISPR/Cas9介导的基因编辑 |
利用微流体技术分离携带添加条形码的编辑基因的单个细胞。随后提取出细胞中的mRNA,并利用携带条形码的引物对它们进行测序 |
分析1000个基因扰动需要4万美元 |
分析1000个基因扰动需要12个小时 |
|
注:*指的是这些估计值是基于Aviv Regev和同事们的未发表的数据得出的。 |
||||
本文系生物谷原创编译整理,欢迎转载!点击 获取授权 。更多资讯请下载生物谷app。
原始出处:
Massively Parallel Perturbations
参考文献:
1.Perturb-Seq: Dissecting Molecular Circuits with Scalable Single-Cell RNA Profiling of Pooled Genetic Screens
Cell, 15 December 2016, doi:10.1016/j.cell.2016.11.038
2.Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq
Cell, 15 December 2016, doi:10.1016/j.cell.2016.11.039
3.A Multiplexed Single-Cell CRISPR Screening Platform Enables Systematic Dissection of the Unfolded Protein Response
Cell, 15 December 2016, doi:10.1016/j.cell.2016.11.048
 基因君官网
基因君官网